El trigo harinero (Triticum aestivum) es alohexaploide con 42 cromosomas (siendo n = 7 para cada juego cromosómico; como son 6, pues 6x/ = 42): procede de la hibridación de tres especies diferentes:
T. urartu (2n = 14): Aportó un juego cromosómico A (7 cromosomas). Extinta.
Sitopsis (2n = 14): Aportó un juego cromosómico B (7 cromosomas). Extinta.
Se creó un híbrido AB de 14 cromosomas, pero como ambos juegos A y B son diferentes, el híbrido no podría reproducirse. En algunos casos, se solucionó el problema de infertilidad, duplicando los cromosomas, de modo que salió un trigo tetraploide AABB (2n = 28), que fue el T. turgidum. De similar forma se originó el T. durum (2n = 28, AACC) por hibridación del T. urartu con Aegilops speltoides.
El alotetraploide T. turgidum se hibridó posteriormente con Aegilops taushii, que aportó el genoma D, que se unió a los gametos del T. turgidum, AB, dando un híbrido ABD, que nuevamente, para evitar los problemas de esterilidad, duplicó sus cromosomas, dando lugar al alohexaploide AABBDD T. aestivum, hace unos 2.000 años.
Actualmente, los trigos más cultivados son el harinero, para panificación, y el duro (7% del cultivo de trigo mundial) para pasta y sémola (cuscús), con alto contenido en gluten (13%), es decir, uno hexaploide y otro tetraploide. Otro trigo hexaploide es el espelta (T. spelta), que aunque contiene también gluten, como todos los trigos, y por tanto, no puede ser consumido por celíacos, contienen menos que los otros dos (sólo un 5%), por lo que puede ser mejor para personas con SGNC (Sensibilidad al Gluten No Celíaca).
Los siguientes cereales contienen gluten: todas las especies de trigo, centeno, cebada y avena. No contiene el maíz.
LOS PLÁTANOS SON TRIPLOIDES, por lo que son estériles y se deben reproducir vegetativamente (asexualmente), lo que pone en peligro sus poblaciones.

Algunos peces, como el carpín dorado, pueden ser poliploides, aunque este fenómeno es mucho más raro en animales que en plantas.
La poliploidía se puede inducir mediante el uso de la colchicina que para la división celular antes de que se produzca la separación cromosómica. Las ventajas puede ser obtener semillas o frutos mayores. Un alopoliploide inducido, creado a finales del siglo XIX, es el Triticale, híbrido del trigo con el centeno.
 HEMOTECA de los islandeses. Colección de DeCODE:
HEMOTECA de los islandeses. Colección de DeCODE:


 Fue encontrado en una roca de EE UU.
Fue encontrado en una roca de EE UU.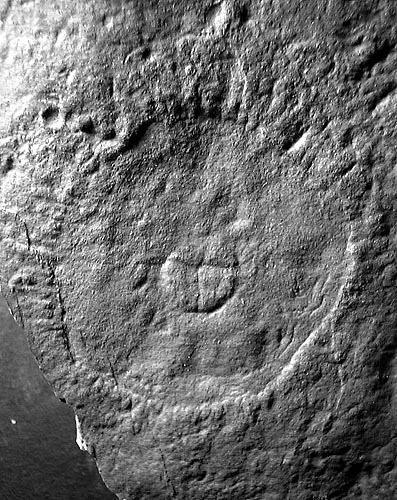 Paleofragmodyctia, encontrado en el sur de Australia, el país de los canguros, koalas y ornitorrincos:
Paleofragmodyctia, encontrado en el sur de Australia, el país de los canguros, koalas y ornitorrincos:

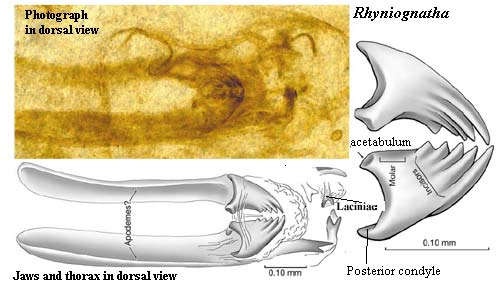 Insecto más antiguo encontrado (400 M.a.)
Insecto más antiguo encontrado (400 M.a.) Acantostega, 370 M.a.
Acantostega, 370 M.a.









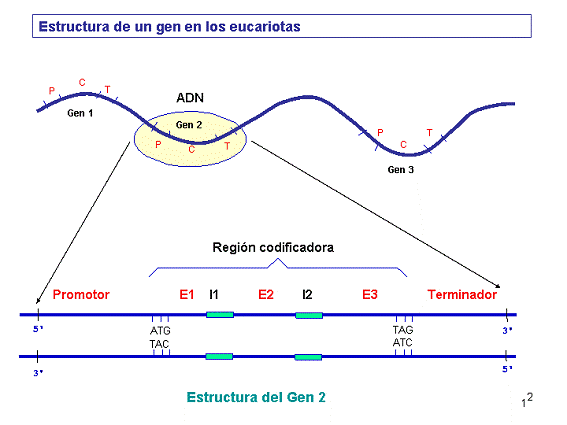 ¿Cuál de las dos cadenas es el gen a transcribir?
¿Cuál de las dos cadenas es el gen a transcribir? Las secuencias 1 y 2 pertenecen a E. coli. La 3 a la levadura.
Las secuencias 1 y 2 pertenecen a E. coli. La 3 a la levadura.

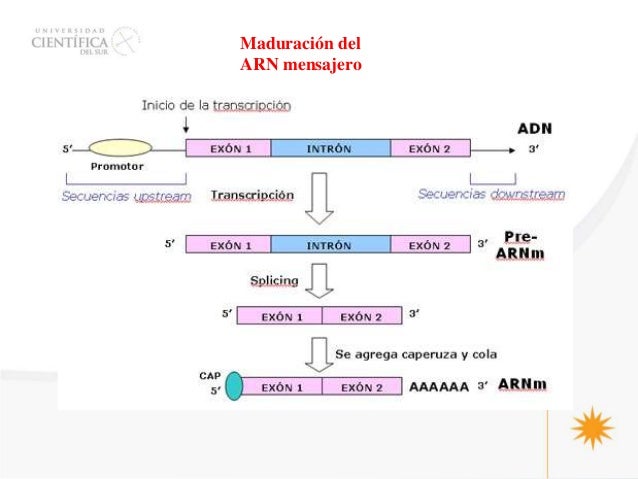
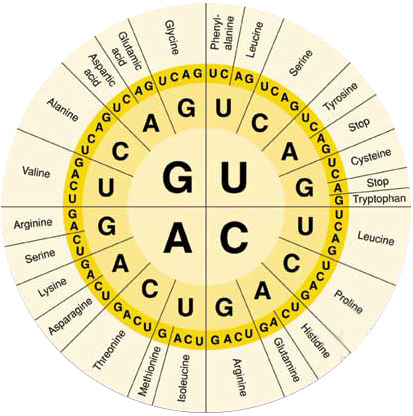


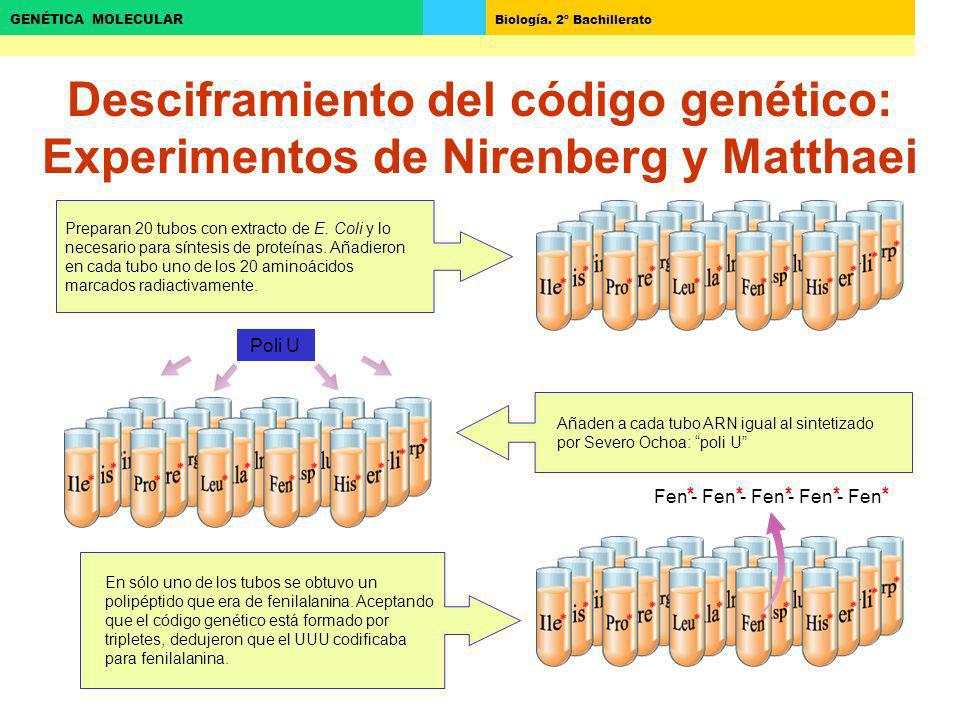


 Ácido homogentísico o melánico (también se acumula en las articulaciones y cartílagos, produciendo su oscurecimiento).
Ácido homogentísico o melánico (también se acumula en las articulaciones y cartílagos, produciendo su oscurecimiento).